■
行列の積の計算は%*%を使う。
> a <- matrix(1:4, 2, 2)
> a
[,1] [,2]
[1,] 1 3
[2,] 2 4
> b <- matrix(0:3, 2, 2)
> b
[,1] [,2]
[1,] 0 2
[2,] 1 3
> a+b
[,1] [,2]
[1,] 1 5
[2,] 3 7
> a%*%b
[,1] [,2]
[1,] 3 11
[2,] 4 16
> a*b #これは要素ごとの積で行列の積ではない
[,1] [,2]
[1,] 0 6
[2,] 2 12リストとは、異なるデータ構造を集めて1つのオブジェクトにしたもの。
リスト形式にしたデータに、リストの要素そのものにアクセスするには の二重括弧を用いる。[ ]の括弧だとベクトルとしてのアクセスを行うことになる。
> x <- list(1:3, 4:6, 7:9) #要素数を揃える必要はない > x [[1]] [1] 1 2 3 [[2]] [1] 4 5 6 [[3]] [1] 7 8 9 > x[[1]] #要素の取り出し [1] 1 2 3 > x[1] #リストの要素番号まで取り出される [[1]] [1] 1 2 3
■
大規模データ,比較的手が届くもののリスト.
- Wikipediaのダンプデータ
- 日本の統計サイト, e-Satマクロなやつ多し.
- Mathematica提供のデータを使う.(Mathematicaのライセンスは必要.)
- AmazonのPublic Data Sets(上記サイトで紹介されていたものの1つ)
- The Free 'Big Data' Sources Everyone Should Know まとめサイト発見.ただし,すでに知っているのも多い.(2015.01)
■
列方向のデータのソートを行う。
input : 999999 37705 55754
output: 37705 55754 999999
BEGIN{
FS = " "
}
{
for(k=1;k<=NF;k++){
id[k] = $k
}
sort(id,NF)
for(k=1;k<=NF;k++){
printf id[k]" "
}
printf"\n"
}
END{
}
function sort(ARRAY,ELEMENTS,tmp,i,j){
for(i=2;i<=ELEMENTS;i++){
for(j=i;(j-1) in ARRAY && ARRAY[j-1] > ARRAY[j];j--){
tmp=ARRAY[j];
ARRAY[j]=ARRAY[j-1];
ARRAY[j-1]=tmp
}
}
return
}
■
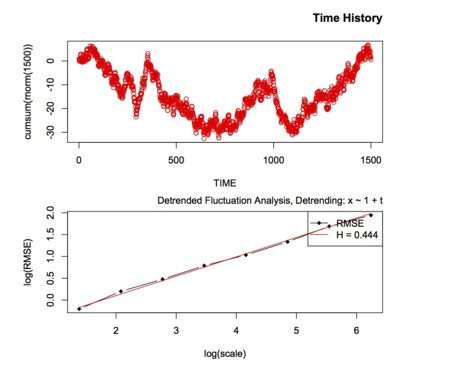
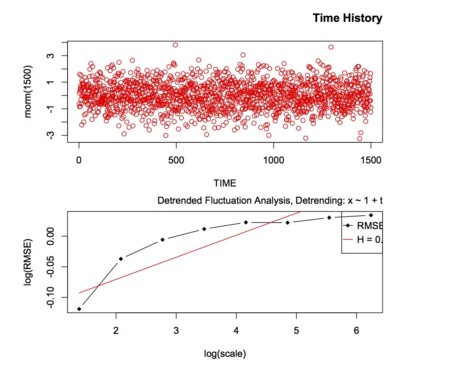
先日読んだ論文で使われていたDFAを試してみようとRのpackageをインストール。
install.packages("fractal", dependencies = TRUE)
library(fractal)
#正規乱数で試して見る
x <- DFA(rnorm(1024))
print(x)
eda.plot(x)
#正規乱数で試して見る,時間窓は1.1倍刻みで,途中の経過を表示
x <- DFA(rnorm(1024),scale.ratio=1.1,verbose=TRUE)
print(x)
eda.plot(x)
#結果のwindow sizeとresidualを取り出す。
attributes(x)
y <- paste(attr(x,"scale"),attr(x,"stat"),sep=",")
write.table(y,"test.csv",append=FALSE,quote=FALSE,row.names=FALSE,col.names=FALSE)
#結果のH指数を取り出す
z <- attr(x,"logfit")
write.table(z$coefficients[2],"test.csv",append=FALSE,quote=FALSE,row.names=FALSE,col.names=FALSE)
#window内を2次式で近似
z <- DFA(rnorm(1024),detrend="poly2",scale.ratio=1.1)
eda.plot(z)
「scale.ratio: the ratio of successive scales. This argument is used as an input to the logScale function. Default: 2.」なので,
scale.ratio=2だと,Scale 6.00000 12.00000 24.00000 48.000 96.0000 192.0000 384.00。
scale.ratio=1.1だと,Scale 6.0000 7.00000 8.0000 9.00000 10.00000 11.0000 12.000 14.0000 15.0000 17.00000 18.00000 20.00000 22.00000 25.00000 27.00000 30.00000 .... てな具合。